Initialising ...
Initialising ...
Initialising ...
Initialising ...
Initialising ...
Initialising ...
Initialising ...
片岡 隆浩*; 首藤 妃奈*; 直江 翔太*; 矢野 準喜*; 神崎 訓枝; 迫田 晃弘; 田中 裕史; 花元 克巳*; 光延 文裕*; 寺東 宏明*; et al.
Journal of Radiation Research (Internet), 62(5), p.861 - 867, 2021/09
被引用回数:5 パーセンタイル:53.7(Biology)Radon inhalation decreases the level of lipid peroxide; this is attributed to the activation of antioxidative functions. This activation contributes to the beneficial effects of radon therapy, but there are no studies on the risks of radon therapy, such as DNA damage. We evaluated the effect of radon inhalation on DNA damage caused by oxidative stress and explored the underlying mechanisms. Mice were exposed to radon inhalation at concentrations of 2 or 20 kBq/m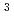 (for 1, 3, or 10 days). For example, the 8-hydroxy-2'-deoxyguanosine levels decreased in the kidneys of mice that inhaled 2 or 20 kBq/m
(for 1, 3, or 10 days). For example, the 8-hydroxy-2'-deoxyguanosine levels decreased in the kidneys of mice that inhaled 2 or 20 kBq/m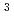 radon for 1, 3, or 10 days. However, the level of Mn-superoxide dismutase (SOD) increased by 15-45% in kidney following radon inhalation. These results suggest that Mn-SOD probably plays an important role in the inhibition of oxidative DNA damage.
radon for 1, 3, or 10 days. However, the level of Mn-superoxide dismutase (SOD) increased by 15-45% in kidney following radon inhalation. These results suggest that Mn-SOD probably plays an important role in the inhibition of oxidative DNA damage.
藤本 浩文*; Pinak, M.; 根本 俊行*; O'Neill, P.*; 久米 悦雄; 斎藤 公明; 前川 秀明*
Journal of Computational Chemistry, 26(8), p.788 - 798, 2005/06
被引用回数:23 パーセンタイル:57.5(Chemistry, Multidisciplinary)電離放射線によるDNAクラスター損傷は修復され難く、細胞のガン化など生体にとって深刻な事態を引き起こす原因の一つと考えられている。DNAクラスター損傷を持つDNA分子には、単独の損傷を持つ分子と比べるとDNA修復酵素が作用し難いことが、生化学的・分子生物学的実験によって示されているが、どのような要因が酵素の作用阻害に関わっているかはいまだ不明である。そこで本研究では、DNAクラスター損傷における酵素の作用阻害の要因を、計算科学的手法を用いて考察した。既報の実験で用いられたDNA分子と同配列となるように、7,8-dihydro-8-oxoguanine(8-oxoG)及びapurinic/apyrimidinic(AP) siteという2つの酸化損傷部位が数塩基はなれた位置に存在する40merのDNA分子を、2損傷部位間の距離を変えて6種類設計し、それぞれに対し分子動力学的(MD)シミュレーションを1nsのオーダーで行った。その結果、損傷部位における分子の屈曲や、損傷塩基と相補鎖上の塩基との相互静電エネルギーの減少など損傷DNA分子に特徴的な構造や性質が観察された。これらの特徴によって修復酵素がDNAに結合できず、したがって修復効率が低下したのではないかと推察される。
Pinak, M.; O'Neill, P.*; 藤本 浩文; 根本 俊行*
AIP Conference Proceedings 708, p.310 - 313, 2004/05
酸化損傷を受けたDNAの構造とエネルギーの変化そしてDNAと修復酵素の複合体形成の動的過程を明らかにするためにDNA突然変異源となる酸化損傷7,8-dihydro-8-oxoguanine(8-oxoG)とその修復酵素human oxoguanine glycosylase 1(hOGG1)を生理的水溶液環境下に配置しmultiple nanosecond moleculardynamics simulationを行った。その結果、DNA-酵素複合体において、N末端のアルギニン324が8-oxoGヌクレオチドのホスホジエステル結合に接近しアミノ酸と損傷部位が化学反応を行えることがわかった。DNA損傷の認識、すなわち修復酵素による損傷部位の認識とDNA-酵素の安定な複合体の形成は、その後の修復プロセスを正常に進めるために必須の条件である。
Pinak, M.
Computational Biology and Chemistry, 27(3), p.431 - 441, 2003/07
被引用回数:9 パーセンタイル:45.16(Biology)30塩基対からなる2種類のDNA(8-オキソグアニン損傷を持つDNA及び持たないDNA)に関して、1ナノ秒の分子動力学シミュレーションを実施した。特に、静電エネルギーに焦点を絞った解析を行った。DNAの静電エネルギー変化は立体構造を崩壊させる重要な要因であると考えられるとともに、損傷の存在を修復酵素に知らせるシグナルとしての役目を果たすことも予想されている。本論文では、これらの機構に関する詳細な解析結果を示す。
Pinak, M.
Journal of Computational Chemistry, 24(7), p.898 - 907, 2003/04
被引用回数:8 パーセンタイル:36.11(Chemistry, Multidisciplinary)突然変異を誘発するDNA酸化損傷である7,8-ジヒドロ-8-オキソグアニン(8-oxoG)と、ヒト修復酵素であるオキソグアニングリコシレース1(hOGG1)とが形成する複合体の分子動力学的シミュレーションを1ナノ秒(ns)行い、DNA-酵素複合体の形成にかかわる動力学的過程の検討を行った。分子動力学的シミュレーション開始後500ピコ秒(ps)でDNA-酵素複合体が形成され、シミュレーションが終了する1ns後まで安定していた。複合体はおもにDNAと酵素のファンデルワールス面の重なり合いによって定義されており、アルギニン313のN末端はヌクレオチドのリン酸ジエステル結合の近傍に位置して、8-oxoGはアミノ酸と損傷と化学反応を可能にしている。また水素結合の切断によって部分的に構造が破壊されたB型DNAが観察された。8-oxoGの5'位のリン酸ジエステル結合は、アルギニン313のN末端に近い位置に移動している。DNAと酵素の近接箇所では水分子を介した水素結合が形成され、複合体の安定性を高めている。正常なDNAを用いた同様の分子系で行ったシミュレーションでは、複合体や水分子を介した水素結合は観察されたなかった。
Pinak, M.; Laaksonen, A.
Molecular Mechanisms for Radiation-Induced Cellular Response and Cancer Development, p.266 - 273, 2003/00
DNAの構造変化とDNA-酵素の複合体形成の動的過程を調べるために、突然変異性の酸化DNA損傷である8-オキソグアニンとその修復酵素であるhOGG1について、DNAのみの系と、DNA-酵素の系についての分子動力学シミュレーションを行った。この結果、損傷部位での水素結合の解離がみられDNAの構造が局所的に崩壊することがわかった。また、8-オキソグアニンの向かい側の隣に位置する相補鎖上のアデニンがDNA二本鎖から外側に飛び出ることがわかった。さらに、8-オキソグアニンのあるDNAとhOGG1とは、MDシミュレーション開始から500psで安定な複合体を形成することがわかった。
Pinak, M.
JAERI-Research 2002-016, 31 Pages, 2002/09
突然変異を誘発するDNA酸化損傷 である8-oxoGについて、損傷DNAの単独存在下、及びヒト修復酵素オキソグアニングリコシレース1 (hOGG1)との共存下で分子動力学的シミュレーションを行い、DNA分子の構造変化とDNA-酵素複合体の形成に関わる動力学的過程の検討を行った。シミュレーションには、中心位に8-oxoG分子を挿入したB型DNAヌクレオチド鎖を用いた。DNAのみのシミュレーションでは、水素結合の切断によって部分的に構造が破壊されたB型DNAが観察され、8-oxoG挿入位から1塩基対分離れた相補鎖側のアデニンがDNA二重鎖からフリップアウトしていた。DNAと修復酵素hOGG1共存下のシミュレーションでは、分子動力学的シミュレーション開始後500psでDNA-酵素複合体が形成され、シミュレーションが終了する1ns後まで安定していた。アルギニン313のN末端は、8-oxoGを持つヌクレオチドのリン酸ジエステル結合に近接し、酵素のアミノ酸とDNA損傷との化学反応を可能にしている。8-oxoGの5'位のリン酸ジエステル結合は、アルギニン313のN末端に近接した位置に移動していた。さらに、DNAと酵素の近接箇所では水分子を介した水素結合が形成され、複合体の安定性を高めていた。
石田 恒
Journal of Biomolecular Structure and Dynamics, 19(5), p.839 - 851, 2002/05
被引用回数:11 パーセンタイル:26.27(Biochemistry & Molecular Biology)DNAのグアニン酸化損傷の影響を調べるために、正常なDNAであるd(CGCGAATTCGCG)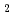 とその酸化的損傷DNAであるd(CGCG
とその酸化的損傷DNAであるd(CGCG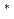 AATTCGCG)(CGCGAATTCGCG),(G
AATTCGCG)(CGCGAATTCGCG),(G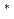 :7,8-dihydro-8-oxoguanine(8-oxoG)の分子動力学シミュレーションを1ナノ秒間、実行した。これは静電相互作用を効率的かつ高精度に計算する高速多重極子展開法(FMM)を用いた高精度シミュレーションである。シミュレーションの結果、8-oxoG付近で正常DNAには見られない新しい水素結合が生成されるのが見つかり、そしてこれが損傷DNAの構造を安定化させていることがわかった。また主鎖角
:7,8-dihydro-8-oxoguanine(8-oxoG)の分子動力学シミュレーションを1ナノ秒間、実行した。これは静電相互作用を効率的かつ高精度に計算する高速多重極子展開法(FMM)を用いた高精度シミュレーションである。シミュレーションの結果、8-oxoG付近で正常DNAには見られない新しい水素結合が生成されるのが見つかり、そしてこれが損傷DNAの構造を安定化させていることがわかった。また主鎖角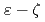 がB
がB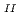 構造,グリコシド結合角
構造,グリコシド結合角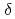 がhigh-
がhigh-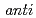 構造,ヘリカルTwistが緩んだ構造をとることもわかった。更に、主成分解析により8-oxoG付近の水和状態の変化が損傷DNAの構造遷移のきっかけとして働いていることが示唆された。以上により、損傷DNA修復酵素は正常DNAと損傷DNAの静的構造及び動的構造の違いを認識していると考えられる。
構造,ヘリカルTwistが緩んだ構造をとることもわかった。更に、主成分解析により8-oxoG付近の水和状態の変化が損傷DNAの構造遷移のきっかけとして働いていることが示唆された。以上により、損傷DNA修復酵素は正常DNAと損傷DNAの静的構造及び動的構造の違いを認識していると考えられる。
石田 恒
Proceedings of the International Conference on Bioinformatics 2002 (CD-ROM), 6 Pages, 2002/02
核酸などの強い静電相互作用を持つ生体高分子の分子動力学には、高速かつ高精度なアルゴリズムが不可欠である。今回、静電相互作用を能率的に取り扱いながら時間ステップを増大するため、高速多重極子展開法(FMM)とマルチ時間ステップ法(MTS)を併用するアルゴリズムを構築した。FMMは計算領域をツリー階層的なセルに分割し、静電相互作用を高速かつ高精度に計算する。なお、このFMMは空間分割法で並列化された。MTSではLiouville演算子の高次展開法を用いてFMMにおけるセルの階層に適用できるようにした。この手法を用いて7,8-dihydro-8-oxoguanine(8-oxoG)を含む8-oxoG DNAの分子動力学を実行した。その結果、8-oxoG付近で主鎖角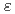 -
-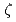 がB
がB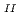 構造,グリコシド結合角
構造,グリコシド結合角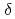 がhigh-
がhigh-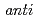 構造,
構造,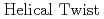 が異常に低い構造をとることがわかった。また、主成分解析により8-oxoG付近の水和構造の変化が8-oxoG DNAの構造遷移のきっかけとして働いていることがわかった。
が異常に低い構造をとることがわかった。また、主成分解析により8-oxoG付近の水和構造の変化が8-oxoG DNAの構造遷移のきっかけとして働いていることがわかった。